イメージング Stellaris アッセイ パート Ⅲ: プロフェッショナルで掲載可能な画像の作成

今回が「イメージングStellarisアッセイ」ブログシリーズの最終回です。 前回までの2つのブログ記事では、顕微鏡ハードウェアと画像取得について説明しました。 この記事では、Raw Z-stack画像を見栄えの良い、掲載可能な画像に変える、簡単なダウンストリーム画像処理についてご説明します。
段階的な概要:
1. Raw画像ファイルを開く
2. 前処理チェックを行う
3. 最大値投影画像を作成する
4. 合成画像を作成する
5. 明るさとコントラストを調整する
6. スケールバーを追加する
7. 最終画像を保存する
さあ、始めましょう。
ImageJのオープンソースの無料配布パッケージである[Fiji Is Just ImageJ](Fiji) 画像処理パッケージを使用します。Fijiは、スタンドアロンのImageJプログラムを拡張して、http://fiji.sc/Downloadsでダウンロードできる多くの便利な機能とプラグインを組み込んでいます。 この記事の手順は、他の画像処理ソフトウェアの大半とも互換性があります。ImageJと
Fijiの詳細については、[Getting Started]のページ (http://fiji.sc/Getting_started) を参照してください。 ImageJチュートリアルページ (http://fiji.sc/Category:Tutorials) には、ImageJの使用方法に関する追加リソースもあります。
Raw画像ファイルを開く
ImageJ を起動した後、 Raw画像ファイルをImageJウィンドウにドラッグすることで、簡単に開くことができます。 まず、前回のブログ記事で取得したMCF7 細胞の細胞増殖バイオマーカーMKI67の60x Z-stackファイルをImageJウィンドウにドラッグします。以前のブログ記事を読んでいない場合、または60Xイメージstackの作成方法を忘れた場合は、 こちらをクリックして確認してください。ファイルをFijiにドラッグすると、[Bio-Formats Import Options] ウィンドウが表示される場合があります。
注:ファイルの種類によっては、Bio-Formats Importerを必要としないものもあります。 Bio-Formats Importerウィンドウが開かない場合は、Bio-Formatsの説明をスキップして、[Pre-Processing check]セクションに進んでください。
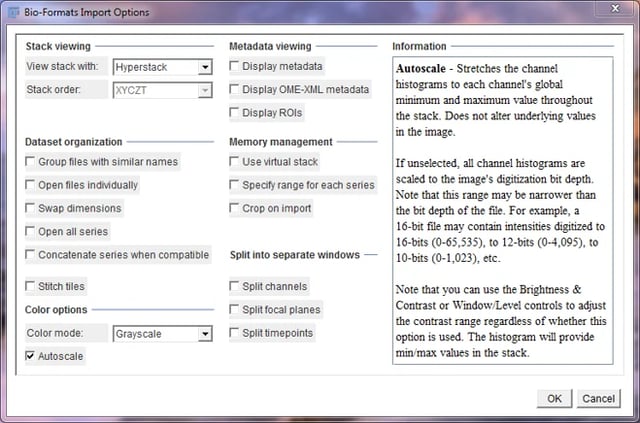 Bio-Format Import Optionsウィンドウ
Bio-Format Import Optionsウィンドウ
[View stack with]のドロップダウンメニューで[Hyperstack] を選択し、[Color mode]ドロップダウンメニューで[Grayscale]を選択してください。[Autoscale]のチェックボックスをオンにすると、画像が見やすくなります。 [OK]を押して続行します。コンピュータの処理速度によっては、ファイルが開くまでに数秒かかる場合があります。ImageJは画像を作業メモリに読み込んでおり、画像が大きい場合は時間がかかることがあります。
ヒント: ファイル展開に度々時間がかかる場合、特にZ-stack内の画像のいくつかを確認したいだけの場合は、[Use virtual stack]のチェックボックスをオンにしてみてください。 ImageJは一度に画像の1スライスのみを読み込むため、画像ファイルを開く速度が大幅に向上します。唯一の欠点は、スライス間の切り替えに非常に時間がかかることです。
前処理チェック
画像が開いたら、スクロールホイールまたは左右の矢印キーを使用して、Z-stack内を移動できます。 Z-stack中の位置は、左上隅または下部のバーで確認できます。
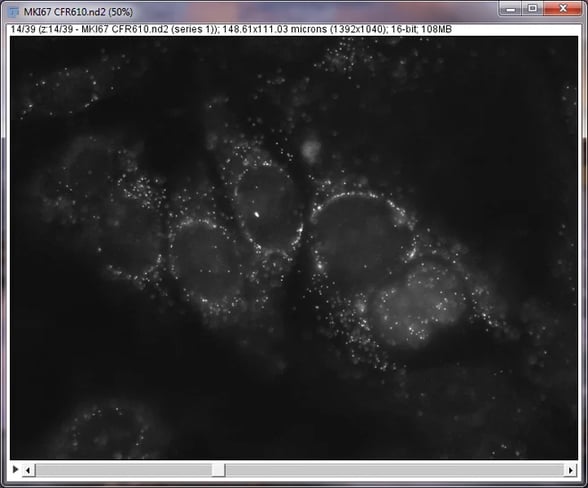 MCF7 細胞の細胞増殖バイオマーカーMKI67からの単一RNA分子を示す単一のz-slice
MCF7 細胞の細胞増殖バイオマーカーMKI67からの単一RNA分子を示す単一のz-slice
この時点で、Z-stackをざっとスクロールして、細胞の端の欠落、 (人為的な)疑似シグナル、XY平面内の大幅なドリフト、浮遊自家蛍光粒子などのエラーを探すことをお勧めします。 このオブジェクトやエラーは、処理後の最終的な画像に含まれる可能性があるため、最終分析で考慮する必要があります。 このエラーに対処する方法については、今後の記事で説明します。
ヒント:Z-stackに何も表示されない場合は、表示範囲が正しく設定されていない可能性があります。Shift+Cを押して、
[Brightness and Contrast] ウィンドウを開きます。 [Auto]をクリックすると、画像が正しい表示値に自動調整されます。 [Auto]ボタンは現在のスライスに合わせて調整されるため、通常はシグナルがあるZ-stackの中央までスクロールすることをお勧めします。 手動調整が必要な場合があります。
最大値投影画像の作成
stackをチェックアウトしたら、Z-stackの最大値投影画像(MaxIP)を作成できます。 MaxIPは、3D Z-stackを2D画像に圧縮するレンダリング方法です。 結果として得られる画像内の各XY位置は、stack全体の中で最も明るいピクセルです。シグナル分子はstack内で最も明るいスポットである必要があるため、これはStellarisの画像に最適です。MaxIPの作成は、内蔵のZ Project ツールを使用して行います。アクセスするには、[Image]>[Stacks]>[Z Project …]をクリックします。
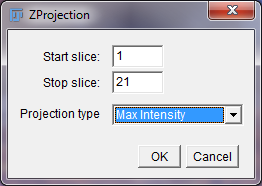
Zプロジェクションオプションウィンドウ
デフォルトでは、[Start slice]ボックスと[Stop slice]ボックスに最大スライス番号と最小スライス番号を設定する必要があります。[Projection type (投影タイプ)]を[Max Intensity(最大値)]に設定し、[OK]をクリックします。
ヒント: stackの両端に余分なスライスが多くある場合は、MaxIPを適切な領域におさめるために、開始slice番号と
停止slice番号、あるいはその両方を変更することをお勧めします。
おめでとうございます!最初のMaxIPを作成しました。掲載できる品質の画像作成に、また一歩近づいています。
次に、取得したDAPI Z-Stackを開いてください。上記と同じ手順に従って画像を開き、DAPI Z-StackのMaxIPを作成します。ImageJがどのウィンドウでMaxIPを実行するかがわかるように、新しく開いたDAPIウィンドウをクリックしてください。
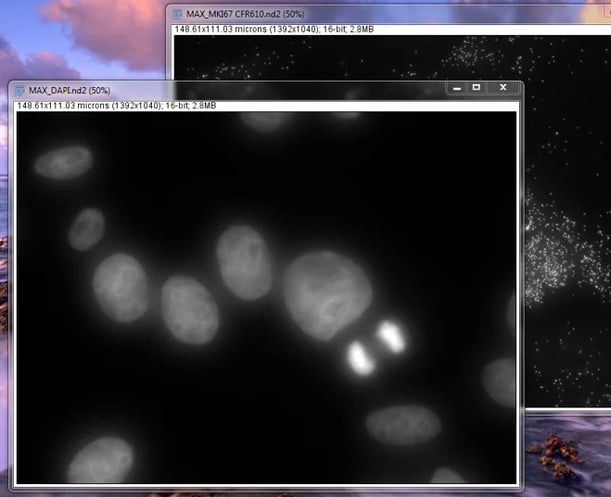 MCF7細胞のDAPIおよびMKI67のMaxIP
MCF7細胞のDAPIおよびMKI67のMaxIP
合成画像の作成
2つのMaxIP画像を開いたら、両者を組み合わせた合成画像を作成します。 核染色と Stellarisシグナルを区別しやすくするために、DAPIチャネルを青色に疑似色付けします。 メニューで[Image]>[Color]>[Merge Channels…]をクリックして実行します。以下のウィンドウが表示されます。
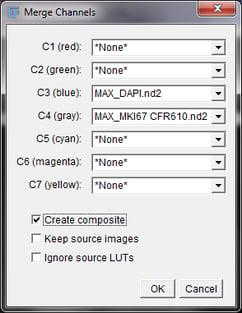
[Merge Channels Options]ウィンドウ
こC3 (青) のドロップダウンメニューで、DAPI画像のタイトルを選択します。次に、C4 (グレー) で、Stellarisの画像のタイトルを選択し、[OK]を押します。 MaxIP画像のタイトルは[MAX_]で始まる必要があります。これにより、DAPIチャンネルが青に見え、Stellarisシグナルが白/グレーに見える合成画像が作成されます。 任意の色の組み合わせを使用できますが、Stellarisプローブセットを1つだけ使用する場合は、青と白が好ましいです。 [OK]をクリックすると、以下のような画像が表示されるはずです。
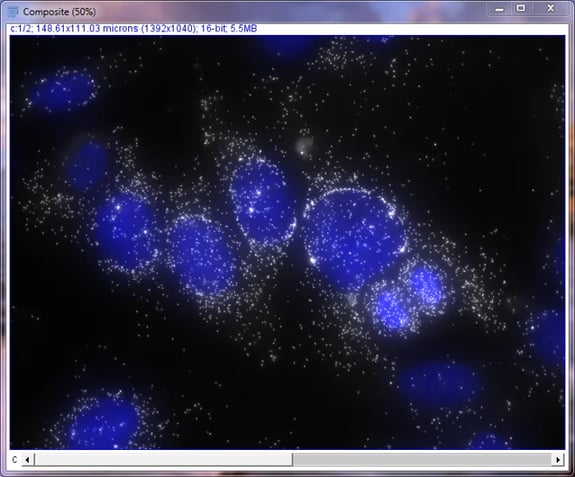
MCF7細胞におけるDAPIとMKI67の合成画像
この画像はすでに見栄えが良いかもしれませんが、画像掲載には明るさの追加とコントラスト調整が必要になります。 次に、いくつか基本的な調整を行います。画像の下部にあるスクロールバーが戻りましたが、フォームが変わることに注意してください。 Z-StackのスクロールホイールではZ平面をスクロールできましたが、このスクロールホイールを使用すると、合成画像内の1色を選択できます。 これにより、元の画像の一方に影響を与えることなく、もう一方の画像に変更を加えることができます。先ほどのZ-Stackと同様に、左右の矢印キーまたはスクロールホイールを使用して、これらの画像を切り替えることができます。また、画像を切り替えると、枠線とヘッダーのテキストの色が変わることにも注意してください。青はブルーチャンネル (DAPI)、黒はグレーチャンネル (MKI67) です。 これを使用して、DAPIとMKI67画像の明るさとコントラストを個別に変更します。
明るさとコントラストを調整する
Shift+Cを押して、明るさとコントラストのウィンドウを開きます。
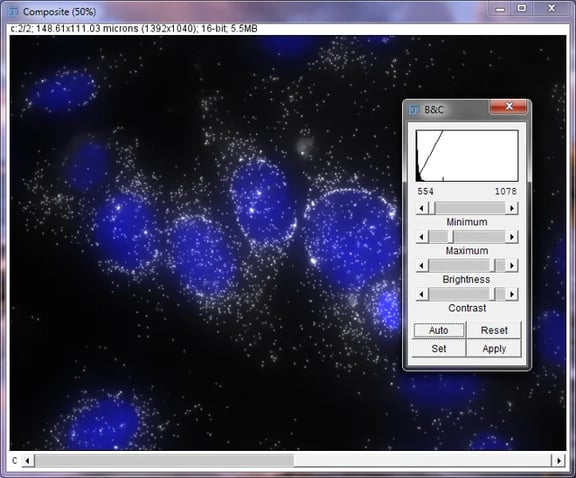
明るさとコントラストウィンドウを備えた MCF7 細胞内のDAPI とMKI67の合成画像
明るさとコントラストのウィンドウは、ヒストグラム、明るさのスライダー、その他の機能ボタンの3つの主要な部分から成り立ちます。ヒストグラムは、画像内のピクセル数を示す棒グラフであり、明るさによって並べ替えられます。左側は暗いピクセル値を表し、右側は明るいピクセル値を表します。このスクリーンショットにはどちらも表示されていませんが、画面の下部に沿って2つの縦の目盛りがあり、表示画像の最小値と最大値を表しています。これらの値はヒストグラムのすぐ下にも表記されます。
明るさのスライダーは、画像の表示方法をコントロールします。 最小値と最大値のバーをスライドさせることで、どの値を黒として表示し、どの値を白とみなすかをコントロールします。 最小値と最大値の間の値は、比例したグレーの階調になります。明るさのスライダーは、最小値と最大値の両方を同時に左右に移動し、コントラストのスライダーは最小値と最大値をさらに離したり近づけたりします。この画像を調整するには、最小スライダーと最大スライダーを使用します。スクロールホイールまたは矢印キーを使用して、画像の MKI67部分を選択します。ヘッダーのテキスト、境界線、およびヒストグラムは黒である必要があります。
まず、画像の最も暗い部分が、モニター上で黒く見えることを確認します。 この多くは、細胞間のスペースです。 この領域はモニター上では黒く表示されるため、最小値を調整する必要はありません。 画像の細胞間のスペースがグレーで表示される場合は、黒になるまで最小スライダーを上(右)にスライドさせてドラッグします。
次に、画像内のシグナルが明るく鮮明に表示されることを確認します。 この画像のシグナルは明るすぎて、光っているように見えます。 バーを右にスライドさせて、この画像のシグナルを暗くして最大値を増やします。 画像内にシグナルが表示されない場合は、最大バーを左にスライドさせてシグナルの明るさを上げてみてください。
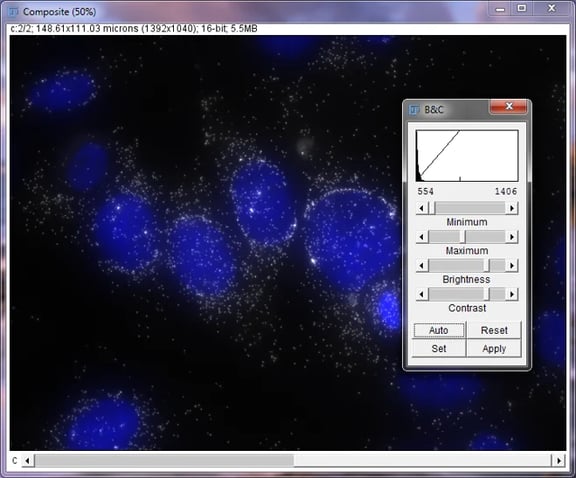
MKI67調整後、明るさとコントラストウィンドウを使用したMCF7 細胞内のDAPIとMKI67の合成画像
次に、スクロールホイールまたは矢印キーを使用してDAPI画像に切り替えます。 その際、必ず [Composite] ウィンドウをクリックしてください。もしクリックしなかった場合、明るさとコントラストの設定を変更することになりますのでご注意ください。 ヘッダーのテキスト、枠線、ヒストグラムの色が青に変わります。 明るさとコントラストのウィンドウに戻ります。 MKI67画像の場合と同じ方法で、DAPIチャンネルを調整します。DAPI画像は核が輝いているように見えることが多いです。 最小値を大きくして輝きを軽減します。
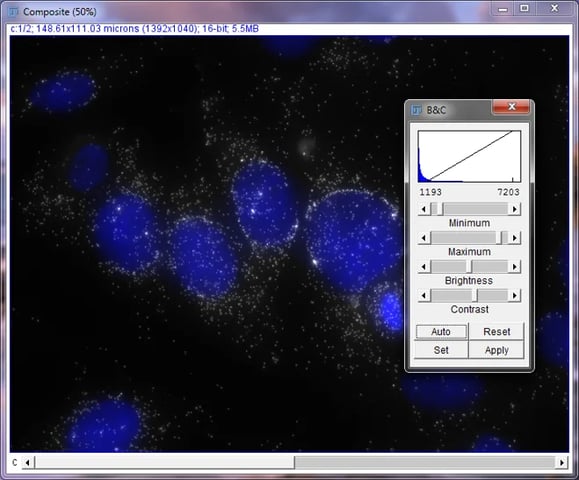 DAPI調整後、[Brightness and Contrast]ウィンドウを使用したMCF7細胞内のDAPIとMKI67の合成画像
DAPI調整後、[Brightness and Contrast]ウィンドウを使用したMCF7細胞内のDAPIとMKI67の合成画像
表示値を自動的に調整したい場合には、[Enhance Contrast]ツールを使用することをお勧めします。 [Enhance Contrast]ツールとは、ほどよく調整された画像を作成する、使いやすいツールで、[Process]メニューの下にあります。 飽和ピクセル値を0から5の間のいずれかに設定します。Stellarisの画像の場合、0から1の間の飽和ピクセル値が適切に機能するケースが多いです。[Normalize]と[Equalize Histogram]のチェックが外れていることを確認してください。
スケールバーの追加
スケールバーを追加するには、内蔵のスケールバーツールを使用します。
[Analyze]>[Tools]>[Scale Bar]をクリックします。 以下のウィンドウが表示されます。
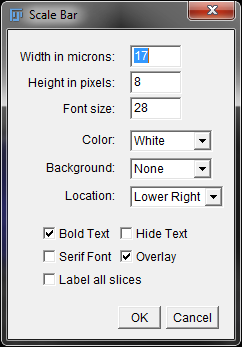 スケールバーオプションウィンドウ
スケールバーオプションウィンドウ
幅をミクロン単位で、適切な数値に設定します。ImageJは、画像のスケーリングとサイズに基づいてデフォルトの数値を選択します。この場合、幅を10ミクロンに変更します。残りの設定はデフォルトのままにしておきます。 必要に応じて、画像の背景や位置を調整してください。 [OK]をクリックして、スケールバーの設定を確認します。
ヒント: スケールバーオプションウィンドウを閉じた後、スケールバーに変更を加える必要がある場合は、スケールバーオプションウィンドウを再度開くことで簡単に変更を加えることができます。 これは、オーバーレイがオンになっている場合にのみ機能します。
最終画像を保存する
適宜画像を調整したら、次は保存の準備に移ります。ファイルは、Raw 16 bit Tiffと8 bit PNGの2つの形式で保存することをお勧めします。Tiffはそれぞれのピクセル値を保存し、将来的に明るさとコントラストをそれぞれ変更する機能がある画像形式です。
[File]メニューを開き、[Save As]にマウスを置きます。[Tiff]を選択します。 保存する場所とファイル名を指定します。 使用した画像はすでに16-bitファイル形式であるため、何も変更する必要はありませんが、もし画像が16-bitでない場合でも、形式変更は行わないでください。
次に、画像を8-bit PNGとして保存します。8-bit画像は、ほとんどのコンピュータで簡単に表示でき、PNGファイル形式は表示したり、他のユーザーと共有したりするのが非常に簡単なファイルです。まず、[Image]>[Type] >[8-bit]に移動して、画像を8-bitに変更します。 次に、[File]>[Save As]>[PNG]の順に選択して、ファイルをPNG として保存します。 希望のファイルの場所を選択し、ファイル名を入力して[Save]をクリックします。
最後に…
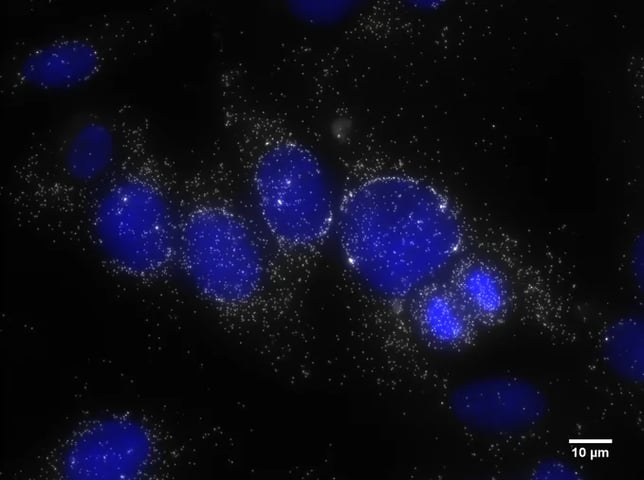
スケールバーを使用したDAPI とMKI67の最終的な合成画像
これで、 Raw画像ファイルから掲載用の画像を作成する基本について、理解が深まったことでしょう。 このコンプリートブログシリーズでは、顕微鏡検査の基礎を紹介しました。この基礎を学ぶことで、皆さんが自信を持って下記の検証を行うことができるように願っています。
・顕微鏡は適切にセットアップされているか
・サンプルの画像化に成功したか
・Raw画像ファイルを掲載できる画像に処理したか
Imaging Stellaris Assaysシリーズがお役に立てば幸いです。 何かご質問やご用命事項がある場合、お気軽にご連絡ください。
Originally published : 2017/08/16 ※最終更新日



