イメージング Stellaris アッセイ パート Ⅱ: 素晴らしい画像を取得する方法

それでは、「Imaging Stellaris Assays」ブログシリーズを続けます。 前回のブログでは、最も重要なコンポーネントである、Stellarisシグナルを検出するために必要な顕微鏡とハードウェア機能の話をしました。 このブログ記事では、細胞を検出して、鮮明で見事な画像を取得するテクニックについて説明します。
 顕微鏡の電源が入っており、必要なコンポーネントがすべて同期され、ソフトウェアに接続されていることを前提として、スライドガラスをホルダーに置き、10 倍の対物レンズをカバーガラスの中心の真下 (倒立スコープを使用している場合) または上 (正立スコープを使用している場合) に移動し、焦点面を探します。これは、接着細胞プロトコルに従って染色されたDAPIチャネルを使用して、細胞の核に焦点を当てるのが最も効果的です。 DAPI(4‘,6’-ジアミジノ-2-フェニルインドール) は、二本鎖DNAのA-Tに富んだ領域に強く結合します。 DNAに結合したこの色素は、最大358nmの光を吸収し、最大461nmで明るく蛍光を発します。 カバースリップの中心の直下または真上に到達したら、DAPIフィルターを選択し、細胞核の検索をスタートします。
顕微鏡の電源が入っており、必要なコンポーネントがすべて同期され、ソフトウェアに接続されていることを前提として、スライドガラスをホルダーに置き、10 倍の対物レンズをカバーガラスの中心の真下 (倒立スコープを使用している場合) または上 (正立スコープを使用している場合) に移動し、焦点面を探します。これは、接着細胞プロトコルに従って染色されたDAPIチャネルを使用して、細胞の核に焦点を当てるのが最も効果的です。 DAPI(4‘,6’-ジアミジノ-2-フェニルインドール) は、二本鎖DNAのA-Tに富んだ領域に強く結合します。 DNAに結合したこの色素は、最大358nmの光を吸収し、最大461nmで明るく蛍光を発します。 カバースリップの中心の直下または真上に到達したら、DAPIフィルターを選択し、細胞核の検索をスタートします。
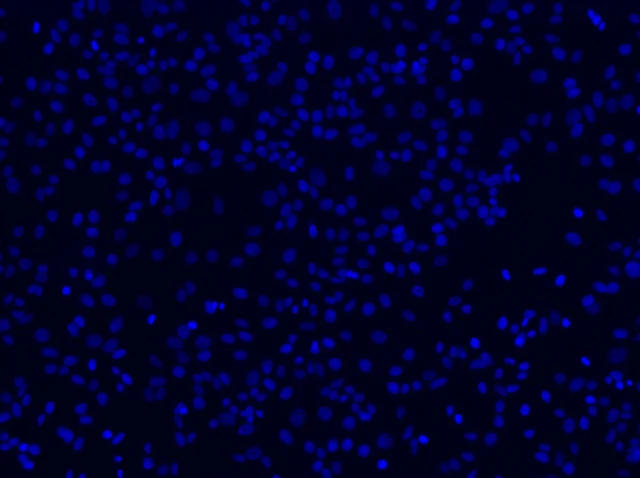 DAPI フィルターを通した細胞核の 10x FOV
DAPI フィルターを通した細胞核の 10x FOV
上記の画像と同等の画像を作成出来たら、対物レンズをスライドから遠ざけ、次に高い対物レンズに回転させます。 対物レンズのみを移動できるように、スライドは固定したままにしてください。 昨今の顕微鏡プラットフォームの多くは、対物レンズを回転するときの最後の位置のZ値を保持するエンコードされたステージが搭載されています。1.3以上の開口数 (NA) の対物レンズに到達するまで、より高倍率の光学系 (20倍、40倍、60倍)に移行していきます。 ご認識の通り、NAはターゲットRNAの単一分子を分解する能力に直接関係しています。
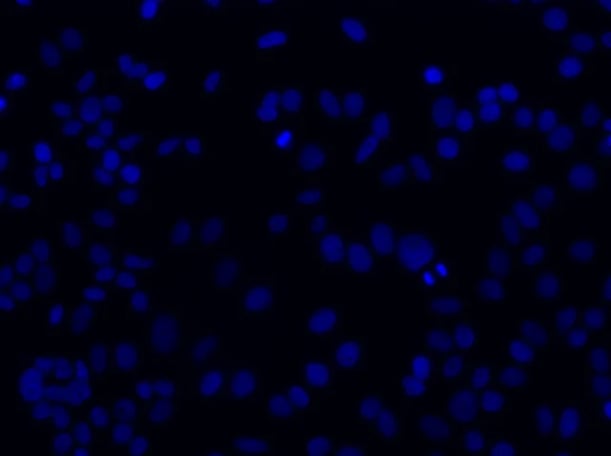 DAPI フィルターを通した細胞核の 20x FOV
DAPI フィルターを通した細胞核の 20x FOV
上記の画像と同等の画像を作成出来たら、60倍の油浸対物レンズに移ります。油浸対物レンズとは、単なる空気ではなく、対物レンズと顕微鏡スライドの間に少量の油が存在する必要があることを意味します。倒立顕微鏡の場合、余分なオイルが対物レンズのバレル内に滴下する可能性があるため、対物レンズの表面ではなくスライド上にオイルを置くことをお勧めします。 油は空気よりも屈折率が高いため、油浸対物レンズは顕微鏡の分解能を高めるために使用されます。 この油浸対物レンズを通って伝播する光により、集光される光の角度が大きくなり、それによってNA値が高くなり、RNAが単一分子に分解されます。
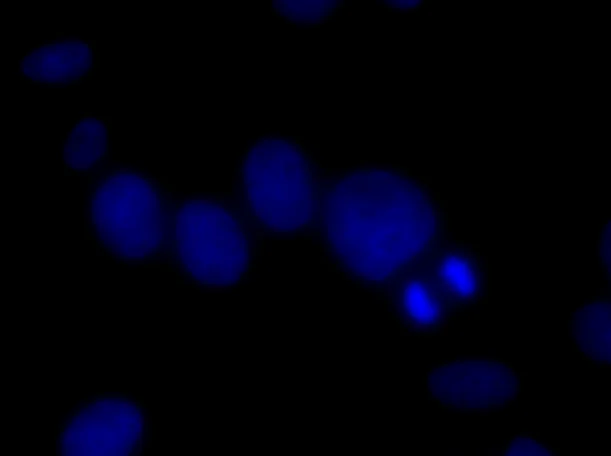 DAPI フィルターを通した細胞核の 60x FOV
DAPI フィルターを通した細胞核の 60x FOV
Stellaris FISHの強力な特長は、別々にラベルされた蛍光プローブの同時検出 (多重化)により、1つまたは複数の遺伝子からのさまざまなRNA バリアントを視覚化できることです。 この異なる蛍光ラベルを画像化するには、狭帯域フィルターを使用して蛍光色素をスペクトル的に分離する手段が必要であることをご認識ください。 DAPIを使用して焦点面を見つけたら、次は、ターゲットRNAに標識された色素分子の検出と励起に適したフィルターを選択します。 露出設定を1秒に調整すると、稼働してサンプルに適切な波長の光を当てる準備が整います。
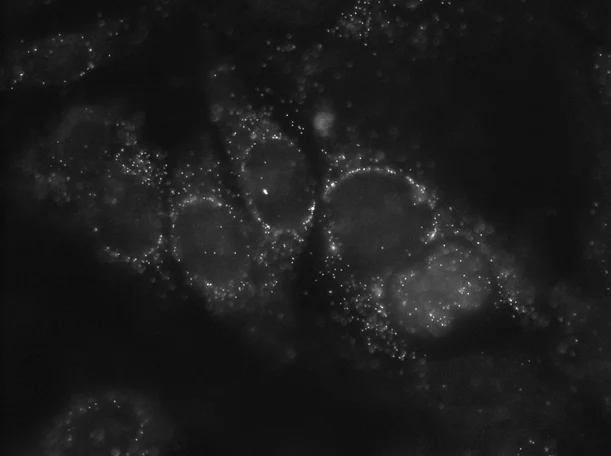 ここでは、MCF7 細胞の細胞増殖バイオマーカー MKI67 からの RNA の単一分子を示すsingle z-sliceに注目します。
ここでは、MCF7 細胞の細胞増殖バイオマーカー MKI67 からの RNA の単一分子を示すsingle z-sliceに注目します。
この画像は、ある時点での単一スナップショットです。 別の言い方をすると、これは単一の焦点面(z-plane)です。
z-planeは一般にz-sliceと呼ばれ、この特定のz-sliceの厚さは0.30ミクロン(μm) です。この単一のz-planeの画像だけでは掲載するには不十分です。その理由は、この単一焦点面の厚さがMCF7細胞の厚さよりも薄いため、細胞のRNAの多くが、焦点から外れたままになるためです。
このアイデアをさらに理解するために、一歩引いて考えてみましょう。 1800年代後半、 Carl Zeiss光学工場の研究責任者Ernst Abbeは、レンズが鮮明な画像を形成するための規則を書き留めました。彼は、作動距離、視野、被写界深度、解像度の、4点の根本的な改善が可能であると書いています。 長年にわたる厳しい教育と訓練を受けた科学者として、あなたはトレードオフの概念、進化の根底にあるメカニズム、そして地球上の生命の存在を強く認識されていることでしょう。 光学分野でもそれは変わりません。 解像度が高くなるほど、視野、被写界深度、作動距離が大幅に損なわれるのです。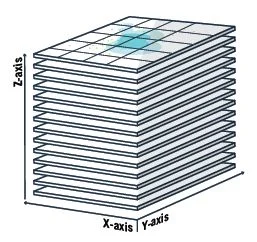 単一のRNA分子に分解できるかどうかは、被写界深度を浅くする、開口数の高い対物レンズを備えているかどうかにかかっています。たとえば、MCF7セルの厚さが平均9µmであるとします。 厚さ1μmの単一の焦点面をキャプチャするということは、細胞の他の8μmで見られるRNA発現を見逃すことを意味します。 細胞の総RNA含有量を理解することは、建物の構造を理解することに似ています。 単一のフロアの設計図だけでは不十分で、各フロアの設計図を編集する必要があります。 細胞の上下に焦点を合わせてイメージングすることで、関連する RNAの発現を見逃すことなく観察できます。 これはz-stackingとして知られる焦点合成技術です。 z-stackingは、対象の細胞中の複数のz-planesをキャプチャしたものです。 細胞内を段階的に「stepping」していくところを想像してみてください。 セル内のステップまたはストライドのサイズは、各z-sliceの厚さに相関します。
単一のRNA分子に分解できるかどうかは、被写界深度を浅くする、開口数の高い対物レンズを備えているかどうかにかかっています。たとえば、MCF7セルの厚さが平均9µmであるとします。 厚さ1μmの単一の焦点面をキャプチャするということは、細胞の他の8μmで見られるRNA発現を見逃すことを意味します。 細胞の総RNA含有量を理解することは、建物の構造を理解することに似ています。 単一のフロアの設計図だけでは不十分で、各フロアの設計図を編集する必要があります。 細胞の上下に焦点を合わせてイメージングすることで、関連する RNAの発現を見逃すことなく観察できます。 これはz-stackingとして知られる焦点合成技術です。 z-stackingは、対象の細胞中の複数のz-planesをキャプチャしたものです。 細胞内を段階的に「stepping」していくところを想像してみてください。 セル内のステップまたはストライドのサイズは、各z-sliceの厚さに相関します。
z-stackingを使用すると、3-Dサンプルボリューム全体をキャプチャできます。
下流処理では、3-Dデータがレンダリングされ、2-D画像に圧縮されます。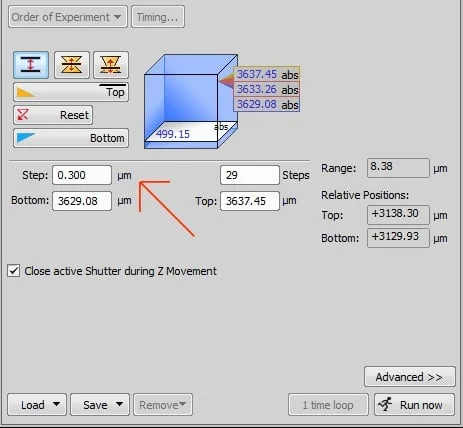
これは、z-stackingとステップサイズのパラメーターを調整できる取得ボックスです。
ここではステップサイズを変更する方法を説明します。 0.25~0.30μmのステップサイズで z-stackを取得することを推奨します。経験豊富な顕微鏡技師は、より大きなステップサイズを使用して取得速度を上げたり、露光量を減らしたりすることがありますが、トレードオフを理解することが常に重要です。
現在、多くの広視野蛍光顕微鏡システムには、z-stackの自動キャプチャを可能にする電動ステージが装備されています。 時間はかかりますが、各焦点面を手動でステップ実行して各z-sliceを個別に取得することにより、z-stackを収集することは可能です。いずれにせよ、最終的にお伝えしたいことは変わりません。研究者として、z-stackingを通じて実験に関連するすべてのデータを取得することが重要です。 この焦点合成技術を通じて焦点深度を再構築し、完全に焦点の合った画像を作成することができます。
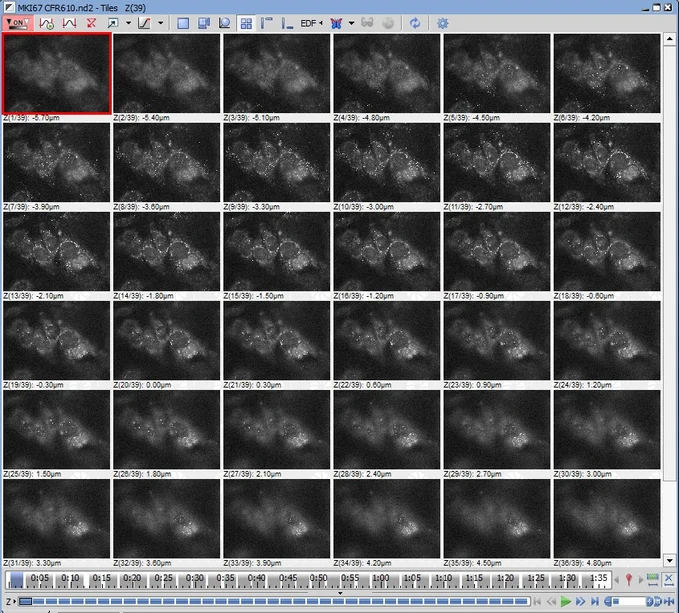 これらは、MKI67の全RNA発現をキャプチャするための完全なz-stackを形成する 36 個の個々のz-sliceです。
これらは、MKI67の全RNA発現をキャプチャするための完全なz-stackを形成する 36 個の個々のz-sliceです。
各z-sliceの厚さは 0.3 ミクロンです。細胞内を進むと、様々なRNAが焦点に入ったり、外れたりします。
ターゲットRNAのz-stackを取得したら、DAPIフィルターに戻すことができます。 同じXY座標とz-stackパラメーターを維持して再取得できますが、今回は視野内のDAPI染色された核の関連する焦点面をすべてキャプチャします。下流の画像処理により、対応する細胞核内に目的の RNAを重ね合わせることができるため、視野が同一であることが重要です。
顕微鏡が適切にセットアップされ、目的のターゲットRNAを正常に画像化することができました!このシリーズが優れた画像を取得するのに役立つだけでなく、顕微鏡の基礎に更なる洞察と理解をもたらすことを願っています。 顕微鏡使用方法は難しく、焦燥感に駆られることもありますが、ブログに記載した内容は最初に従うべき基本的な作業です。この作業を実施すれば、観察がよりエキサイティングになることをお約束します。 このブログシリーズの最後の記事では、下流の画像処理について説明しますので、どうぞご期待ください。
Originally published : 2017/08/16 ※最終更新日



